Introducción a tidyr: Datos ordenados en R
José Antonio Ortega
Universidad de Salamanca6 de febrero de 2016
- Instalación y carga de paquetes
- Lectura de datos
- De formato ancho a formato largo: la función
gather - Separando variable y fecha con
separate - Código más simple con el operador después
%>% - De formato largo a formato (más) ancho: la función
spread - Gráficos con los datos en forma ordenada
- Código completo
- Hemos visto:
- Referencias básicas
- Referencias generales sobre R
- Referencias citadas
- Notas
Tidyr es un paquete de R que permite ordenar datos “sucios” para obtener objetos de datos en R (tbl_df, muy parecidos adata.frame) en formato ordenado (tidy). La idea de los datos ordenados es que queden organizados del siguiente modo (Wickham, 2014):
- Cada variable está en una columna
- Cada observación está en una fila
En esta introducción a tidyr vamos a hacer una aplicación no trivial que consiste en lo siguiente:
- Leer desde internet un fichero excel que contiene datos correspondientes a variables demográficas de las distintas CCAA españolas en distintos años.
- Transformar los datos a un formato ordenado utilizando el paquete
tidyr. - Una vez que están los datos en formato ordenado, visualizar los datos con un gráfico.
El problema de los datos originales se ve en nombres de columnas en excel como “Nacimientos 2004”. Esto no es formato ordenado porque “Nacimientos” corresponde a la variable, mientras que 2004 es parte de la información que identifica la observación, y debe corresponder a una variable que se llame Fecha o Año. Las observaciones quedarán entonces identificadas por la combinación de territorio y fecha. Transformar los datos no es trivial ya que hay numerosas variables, cada una de ellas observada en varios años. Tenemos tres fases lógicas que se corresponden con tres funciones de tidyr:
- La función
gatherpara pasar de un formato “ancho” a un formato “largo”: cada fila corresponderá a una única combinación de variable y observación. - La función
separatepara pasar de las etiquetas de columna iniciales, que mezclaban observación y variable, a dos columnas diferentes, una con la variable, otra con la fecha. Veremos una forma de escribir código alternativa que simplifica mucho la escritura y la comprensión: las llamadas pipas%>%. Compararemos el código con ellas y sin ellas. - La función
spreadpara que cada variable diferente tenga su propia columna y cada observación su propia fila.
Por último, procedemos a mostrar cómo los datos ordenados nos permiten ya el análisis de los datos. Vamos a graficarlos utilizando funciones del paquete lattice, pero también sería ya posible estimar modelos econométricos, en este caso para datos de panel: tenemos un panel de datos puesto que tenemos observaciones para una serie de variables en distintos momentos.
Instalación y carga de paquetes
En R, para poder utilizar un paquete tiene que estar instalado. Si no está instalado dará un mensaje de error. En ese caso habrá que instalarlo, un proceso sencillo desde un ordenador con acceso a internet. Cada paquete se carga en R mediante la función library. El siguiente código carga los paquetes que nosotros emplearemos en este ejemplo. Lo que aparece a partir de la almohadilla, #, son comentarios, no código, para que sepáis que hace cada paquete:
library(dplyr) # Manipulación de data.frames
library(tidyr) # Datos ordenados
library(readxl) # Leer ficheros excel
library(lattice) # Gráficossi hemos obtenido algún mensaje de error (no de advertencia, warning, que avisan de algo pero no impiden la carga), tendremos que instalar los paquetes que corresponda con la función install.packages. lattice está siempre porque forma parte del R básico. Para instalar los paquetes tenéis que ejecutar lo siguiente (sólo con los paquetes que os dieron error).
install.packages("dplyr") # Manipulación de data.frames
install.packages("tidyr") # Datos ordenados
install.packages("readxl") # Leer ficheros excelAl ejecutar install.packages la primera vez nos pide seleccionar desde que repositorio bajamos los datos. Selecciona el que quieras y deja a R instalar los paquetes. La instalación sólo hay que hacerla la primera vez que se utiliza un paquete. R descarga los ficheros necesarios en el lugar adecuado dejándolos preparados para cuando queramos cargar la libreria. Una vez instalados los paquetes, entonces, tenemos que cargarlos con las funciones library que nos dieron problemas anteriormente. Ahora ya deben funcionar. Cargar un paquete en R sólo quiere decir que se incluye la carpeta en que está el paquete en las carpetas en las que R busca los objetos, incluídas las funciones, cuando se le pide. Podemos comprobarlo con la función search que lista las carpetas en las que R busca los datos y que ahora deben incluir dplyr, tidyr y readxl:
search()Lectura de datos
Los datos que queremos cargar en R están disponibles en http://internotes.cajaespana.es/pubweb/decyle.nsf/. Desde esta página los podemos seleccionar con el cursor y cargarlos en nuestro ordenador. Se trata de los datos por comunidades autónomas, de Demografía > Movimiento natural de la población. Queda identificado por “Regiones_Demografia_MNP”. Pero no es necesario hacerlo manualmente. R permite cargar directamente desde la página web el fichero xls, guardándolo en la carpeta de trabajo. Para ello utilizamos la función download.file que tiene como primer argumento la url, y como segundo el nombre del fichero de destino. Desde ahí lo podemos leer y asignarlo a un matriz de datos de R. Esto lo hace la función read_excel:
download.file("http://internotes.cajaespana.es/pubweb/decyle.nsf/E27E3D29593A8FC1C12578720023B795/$File/Regiones_Demografia_MNP.xls",
destfile = "Regiones_Demografia_MNP.xls", mode="wb")
MNPorig = read_excel("Regiones_Demografia_MNP.xls",skip=10)En la función read_excel hemos tenido que identificar el fichero, y cambiar sólo uno de los argumentos por defecto con skip=10. Con ello le estamos diciendo que se salte las primeras 10 líneas del fichero excel, empezando los datos en la 11. Para ver los argumentos que admite la función read_excel, o cualquier otra función, tenemos que escribir
args(read_excel)## function (path, sheet = 1, col_names = TRUE, col_types = NULL,
## na = "", skip = 0)
## NULLo si queremos la página de ayuda
help(read_excel)Comprobamos que por defecto read_excel carga la primera hoja del fichero, asume que la primera fila son los nombres de las columnas, y que el tipo de datos de cada columna debe identificarlo la propia función. De este modo ya tenemos cargados los datos en R, en un objeto que se llama MNP. Para ver la estructura del objeto podemos hacer
str(MNPorig)que nos informa sobre la clase del objeto (derivado de las tablas de datos en R, data.frame), de sus dimensiones (17 observaciones y 93 variables), de los nombres de cada variable, su tipo, y los primeros valores. Vemos que las dos primeras variables,idTerritorio y Territorio son de tipo character (chr) e identifican la CCAA. Las siguientes 91 columnas son numéricas y corresponden a las combinaciones de variable-año. Cada variable está observada entre los años 2004 y 2010. Si queremos abrir el objeto y examinarlo un poco más lo podemos hacer con
View(MNPorig)Recuerda cerrar la ventana que se ha abierto cuando no la necesites más.
De formato ancho a formato largo: la función gather
El objeto MNPorig es un ejemplo de datos en formato ancho. Incluye muchas columnas que no se corresponden con variables. Para pasar a datos ordenados, lo primero que haremos es pasarlo a un formato largo. Esto lo hacemos con la función gather que nos pide como argumentos principales:
data: El objeto de datos, en nuestro casoMNPorigkey: El nombre que asignamos a la columna que incluya las etiquetas de las columnas en formato ancho. La podemos llamarcolumnavalue: El nombre que asignamos a la columna que incluye los valores de dichas columnas, los numeros. La podemos llamarvalor- …: Esto se refiere a la especificación de las columnas que queremos que pasen a estar en formato largo, con una fila distinta para cada valor de la columna. En nuestro caso son todas las columnas mixtas de observación-variable. Desde la 3 a la 93. Hay distintas formas para identificar estas columnas: por nombre, por posición, todas las variables entre dos determinadas, por exclusión. En nuestro caso lo más sencillo es excluir a las columnas 1 y 2, que son las únicas “correctas” de nuestros datos. Se excluyen espeficándolas precedidas por el signo “-”. Para obtener nuestro nuevo objeto de datos hacemos
MNPlargo=gather(MNPorig,columna,valor,-1,-2)Podemos comprobar con dim(MNPlargo) que el nuevo objeto tiene por dimensiones 1547 filas y 4 columnas. Para ver las primeras filas podemos hacer
head(MNPlargo)## Source: local data frame [6 x 4]
##
## idTerritorio Territorio columna valor
## (chr) (chr) (chr) (dbl)
## 1 CCA01 Andalucía Nacimientos 2004 89022
## 2 CCA02 Aragón Nacimientos 2004 11458
## 3 CCA03 Asturias (Principado de) Nacimientos 2004 7218
## 4 CCA04 Balears (Illes) Nacimientos 2004 10792
## 5 CCA05 Canarias Nacimientos 2004 19207
## 6 CCA06 Cantabria Nacimientos 2004 5060Vemos que el formato es ahora muy diferente: en lugar de muchas columnas tenemos muchas filas. Cada fila tiene un único dato. En cuanto a los formatos de las columnas, las tres primeras siguen siendo (chr), y valor es una columna numérica con doble precisión ((dbl)). Si inspeccionamos con atención veremos que la columna columna presenta algunos problemas. Podemos verlo con
unique(MNPlargo$columna)donde la notación objeto$col selecciona la variable col del data.frame o lista objeto. La función unique presenta los valores diferentes que toma la variable, en este caso 91. Vemos que, en efecto, algunas etiquetas presentan caracteres leidos erróneamente (acentos y tanto por mil). Este problema se debe a que read_excel no determinó bien el formato del texto. La solución es sencilla:
Encoding(MNPlargo$columna)="UTF-8"Podéis comprobar con unique(MNPlargo$columna) que el problema ha sido resuelto y los textos son los que queríamos. El uso de Encoding ha permitido a R saber que el texto estaba realmente escrito en formato [UTF-8] (https://es.wikipedia.org/wiki/UTF-8).
MNPorig = read_excel("Regiones_Demografia_MNP.xls",skip=10)
MNPlargo = gather(MNPorig,columna,valor,-1,-2)
Encoding(MNPlargo$columna) = "UTF-8"Separando variable y fecha con separate
Para poder separar variables y observaciones, tenemos que partir la columna columna en sus dos componentes. Para ello tidyrtiene la función separate que toma los siguientes argumentos
args(separate)## function (data, col, into, sep = "[^[:alnum:]]+", remove = TRUE,
## convert = FALSE, extra = "warn", fill = "warn", ...)
## NULLdata: El objeto de datos. En nuestro casoMNPlargo.col: La columna que queremos separar en dos o más partes. En nuestro caso,columna. Hay que escribir directamente el nombre de la variable, sin ponerla entre comillas. Existe otra función si queremos especificar la columna como carácter, entre comillas: la funciónseparate_.into: Los nombres de las nuevas columnas que se crean. En nuestro caso podemos llamarVariablea la parte con el nombre de la variable yFechaa la que contiene el año. Para ello hay que especificarlo comoc("Variable","Fecha").sep: Especifica por dónde separar la columna original. Si es un número, es la posición en que separar. En nuestro caso una primera opción parecería ser separar donde haya un espacio. Eso funcionaría con los nombres sencillos, como “Nacimientos 2004”, pero falla con nombres largos como “Migracion Exterior Inmigración 2004” que tienen más de un espacio. La solución más sencilla, aunque requerirá alguna manipulación más, es utilizar ” 2″ como separador. Luego tendremos que sumar 2000 a la fecha.remove: Por defecto es cierto. Controla si la variablecoles reemplazada por las nuevas, o si se mantienen todas. Nos va bien la opción por defecto.convert: Si deben tratar de convertirse las variables nuevas a su tipo en caso de que sean numéricas o lógicas. En nuestro caso, especificando TRUE podemos conseguir que la variableFechase transforme a numérica. Nos interesa. Como por defecto es FALSE, tenemos que especificarconvert=TRUE.
En definitiva, el código para generar las variables separadas, sustituyendo el objeto MNPlargo por otro del mismo nombre, es:
MNPlargo=separate(MNPlargo,columna,c("Variable","Fecha"),sep=" 2",convert=TRUE)Podemos comprobar que el nuevo objeto es como queríamos:
head(MNPlargo)## Source: local data frame [6 x 5]
##
## idTerritorio Territorio Variable Fecha valor
## (chr) (chr) (chr) (int) (dbl)
## 1 CCA01 Andalucía Nacimientos 4 89022
## 2 CCA02 Aragón Nacimientos 4 11458
## 3 CCA03 Asturias (Principado de) Nacimientos 4 7218
## 4 CCA04 Balears (Illes) Nacimientos 4 10792
## 5 CCA05 Canarias Nacimientos 4 19207
## 6 CCA06 Cantabria Nacimientos 4 5060Por último, tenemos que mutar la columna Fecha sumando 2000. Para ello empleamos la función mutate del paquete dplyr que toma como argumentos el objeto de datos, y las transformaciones en las columnas separadas por comas:
MNPlargo = mutate(MNPlargo, Fecha = Fecha+2000)Código más simple con el operador después %>%
Podemos recopilar de nuevo el código que hemos utilizado para definir y transformar MNPlargo:
MNPorig = read_excel("Regiones_Demografia_MNP.xls",skip=10)
MNPlargo = gather(MNPorig,columna,valor,-1,-2)
Encoding(MNPlargo$columna) = "UTF-8"
MNPlargo = separate(MNPlargo,columna,c("Variable","Fecha"),sep=" 2",convert=TRUE)
MNPlargo = mutate(MNPlargo, Fecha = Fecha+2000)Vemos que, a menudo, vamos manipulando objetos intermedios que no nos interesa guardar. En nuestro caso lo hacemos 4 veces con MNPlargo. Para simplicar el código es muy útil el operador %>%, denominado pipe y cuyos creadores (Bache and Wickham, 2016) sugieren que se lea como entonces o después. El paquete dplyr lo incorpora. La idea del operador después es que lo que aparece a la izquierda es el primer argumento de la función de la derecha, de modo que se pueden acumular operaciones leyendo de izquierda a derecha. Es decir: las tres expresiones siguientes son equivalentes:
g(f(x,y))
z=f(x,y)
g(z)
x %>% f(y) %>% g()Como vemos la más compacta, y posiblemente clara, sea la última. Si utilizamos RStudio para trabajar en R, existe un atajo para escribir el operador más rápido: la secuencia CTRL+MAYUS+M. En nuestro caso, un código exactamente equivalente al que hemos generado con el operador después es el siguiente:
MNPorig = read_excel("Regiones_Demografia_MNP.xls",skip=10)
MNPlargo = MNPorig %>% gather(columna,valor,-1,-2)
Encoding(MNPlargo$columna) = "UTF-8"
MNPlargo = MNPlargo %>%
separate(columna,c("Variable","Fecha"),sep=" 2",convert=TRUE) %>%
mutate(Fecha = Fecha+2000)Vemos que el operador después describe lo que hacemos: cogemos los datos en MNPlargo, *después separamos columna en variable y fecha, y, por último, entonces sumamos 2000 a la fecha, que habíamos truncado quitando el 2. No siempre es útil/fácil emplear el operador %>%. Un ejemplo es la asignación que hacemos declarando la codificación de columna.
De formato largo a formato (más) ancho: la función spread
Ahora tenemos unos datos muy largos pero que no están ordenados todavía: tenemos datos que corresponden a la misma observación definida por el año y el territorio que están en distintas filas. Para ponerlos todos en la misma fila debemos emplear la función spread del paquete tidyr. Estos son los argumentos:
data: El objeto de datos, en nuestro casoMNPlargo. Si utilizamos el operador entonces el objeto de datos debe aparecer a la izquierda.key: El nombre de la columna que queremos pasar de formato largo a formato ancho, en nuestro casoVariable, que queremos que pase a definir columnas.value: El nombre de la columna con los valores correspondientes akey. En nuestro casovalor.
Es decir, para conseguir tener los datos en formato ordenado tenemos que ejecutar lo siguiente:
MNP = MNPlargo %>% spread(Variable,valor)
MNP## Source: local data frame [119 x 16] ## ## idTerritorio Territorio Fecha Crecimiento Vegetativo Defunciones ## (chr) (chr) (dbl) (dbl) (dbl) ## 1 CCA01 Andalucía 2004 25986 63036 ## 2 CCA01 Andalucía 2005 25903 65904 ## 3 CCA01 Andalucía 2006 32876 62428 ## 4 CCA01 Andalucía 2007 30838 65224 ## 5 CCA01 Andalucía 2008 34710 65583 ## 6 CCA01 Andalucía 2009 29886 64730 ## 7 CCA01 Andalucía 2010 27730 64471 ## 8 CCA02 Aragón 2004 -1717 13175 ## 9 CCA02 Aragón 2005 -2054 13682 ## 10 CCA02 Aragón 2006 -709 12989 ## .. ... ... ... ... ... ## Variables not shown: Matrimonios (dbl), Migracion Exterior Emigraciones ## (dbl), Migracion Exterior Inmigración (dbl), Migracion Interior ## Emigraciones (dbl), Migracion Interior Inmigración (dbl), Nacimientos ## (dbl), Saldo Exterior (dbl), Saldo Interior (dbl), Tasa Bruta de ## Mortalidad (‰) (dbl), Tasa Bruta de Natalidad (‰) (dbl), Tasa Bruta de ## Nupcialidad (‰) (dbl)
Como vemos, ya lo hemos conseguido. Tenemos un objeto de datos con 119 combinaciones distintas de territorio y fecha que definen cada observación y con 16 variables distintas. Los nombres de las variables los podemos obtener así:
names(MNP)## [1] "idTerritorio" "Territorio" ## [3] "Fecha" "Crecimiento Vegetativo" ## [5] "Defunciones" "Matrimonios" ## [7] "Migracion Exterior Emigraciones" "Migracion Exterior Inmigración" ## [9] "Migracion Interior Emigraciones" "Migracion Interior Inmigración" ## [11] "Nacimientos" "Saldo Exterior" ## [13] "Saldo Interior" "Tasa Bruta de Mortalidad (‰)" ## [15] "Tasa Bruta de Natalidad (‰)" "Tasa Bruta de Nupcialidad (‰)"
Las etiquetas son descriptivas y nos permiten saber a qué corresponden las variables, pero presentan algunos problemas para la manipulación en R como veremos. Algunos nombres de variables tienen espacios y caracteres especiales como el tanto por mil. Aunque sea más claro, no son nombres estándar de variable en R. Es mejor, en la medida de lo posible evitar espacios en el texto (una solución típica es el guión bajo: “Crecimiento_Vegetativo”) y los caracteres especiales.
Gráficos con los datos en forma ordenada
Vamos ahora a hacer una representación gráfica de algunas de las variables. Esto os mostrará lo sencillo que es hacer gráficos complejos con R una vez que los datos tienen la forma adecuada. También es el momento para introducir la notación de fórmulas en R, que nos acompañará todo el curso. En distintos contextos, tanto cuando hablemos de estimación de modelos como para hacer un gráfico, tenemos la idea de separar variables en dos categorías: la (o las) que nos interesa estudiar, que llamaremos variable/s y, y las que sirven para explicarla o encuadrarla, que llamaremos variable o variable x. Las fórmulas de R separan las variables y y x mediante la tilde ~ 1 Estos son ejemplos de fórmulas
y ~ x
y ~ x1 + x2
y1 + y2 ~ x
y1 + y2 ~ x1 + x2
y1 + y2 ~ x | zComo vemos, dependiendo de la aplicación, puede haber una o más de una variable x, una o mas de una variable y. En concreto, cuando hagamos gráficos las variables y son las que queremos que aparezcan en el gráfico en el eje de las y, y la variable x (única en este contexto), la que aparece en el eje de las x. La última fórmula incluye la barra vertical | 2, que en el contexto de los gráficoslattice significa que queremos hacer el análisis condicionando por la variable z. Esto se hace definiendo para cada valor de z un gráfico distinto. Vamos a aplicarlo para representar gráficamente los nacimientos y las defunciones a lo largo del tiempo en las distintas CCAA. La fórmula que describe el gráfico será
Nacimientos + Defunciones ~ Fecha | TerritorioLa función que utilizaremos para los gráficos, xyplot, corresponde al paquete lattice 3
xyplot(Nacimientos+Defunciones~Fecha|Territorio,
data=MNP,auto.key=TRUE)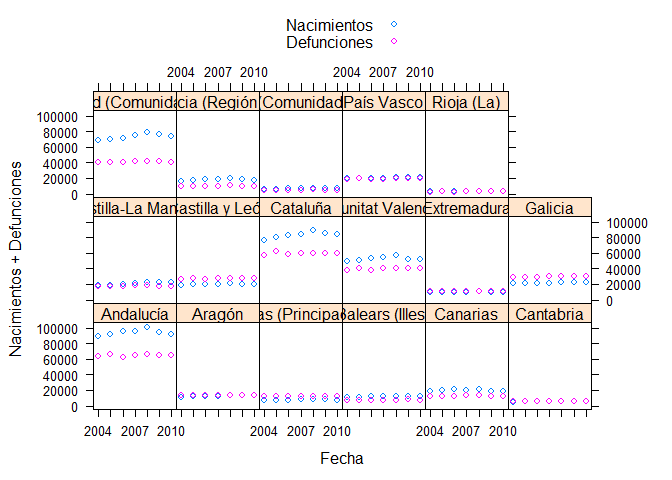
Como primer gráfico no está mal, pero hay varias cosas que podemos cambiar:
- La escala de las variables depende de la población correspondiente. Para eliminar o reducir el efecto de la población total podemos representar las tasas brutas en vez del número de nacimientos, o podemos tomar logaritmos de las variables en el eje y. Probaremos primero esta alternativa, después la segunda.
- No está mal que aparezcan los puntos, que es la opción por defecto, pero podría leerse más facil añadiende líneas que unan los puntos y una rejilla de coordenadas.
xyplotpermite controlar todas las características del gráfico aunque a veces no sea muy sencillo el hacerlo. Podéis hacerhelp(xyplot)(? xyplotes equivalente) o consultar, por ejemplo,http://oscarperpinan.github.io/intro/graficos.html. En particular, añadiendo el argumentotype=c("p","l","g")tenemos puntos, líneas y rejilla. - La leyenda puede quedar mejor debajo del gráfico, controlamos la situación con la opción
auto.key=list(space="bottom") - No se ven completos los nombres de las CCAA, podemos hacer el texto más pequeño (con
cex) o el gráfico más grande (o ambos).
El gráfico queda entonces así
xyplot(log(Nacimientos)+log(Defunciones)~Fecha|Territorio,
data=MNP,auto.key=list(space="bottom"),type=c("p","l","g"),
par.strip.text=list(cex=0.6))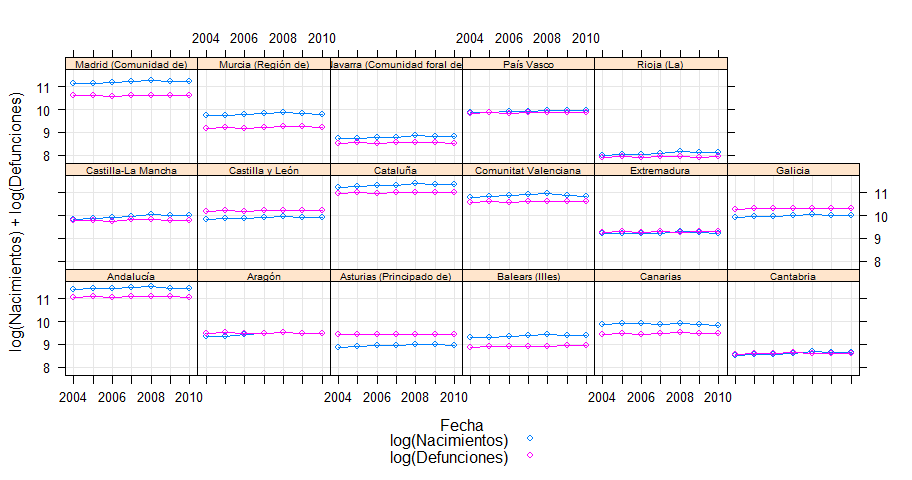
En el gráfico se puede seguir apreciando qué provincias son mayores, pero ya el efecto tamaño no domina el gráfico. También se ve que hay diferentes situaciones respecto a la relación entre nacimientos y defunciones: hay crecimiento natural positivo en comunidades como Madrid o Murcia, crecimiento natural negativo en Castilla y León, Galicia o Asturias. El principal problema para hacer el gráfico de las tasas brutas son los nombres no estándar de las variables, con espacios y caracteres especiales. Estas variables se pueden meter en fórmulas lattice entre acentos graves (`), como en este ejemplo:
xyplot(`Tasa Bruta de Natalidad (‰)` + `Tasa Bruta de Mortalidad (‰)` ~ Fecha | Territorio,
data=MNP,auto.key=list(space="bottom"),type=c("p","l","g"),
par.strip.text=list(cex=0.6))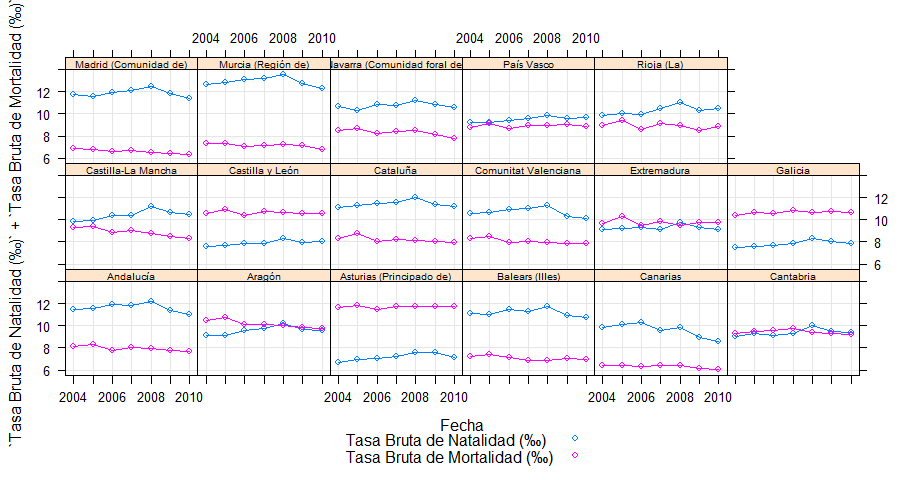
Este gráfico es más claro si cabe. Se ven las grandes diferencias en las tasas brutas de natalidad entre regiones como Madrid o Murcia, en torno a 12‰, y regiones como Asturias, más cercano a 6‰. Por último, podemos ver todos los objetos que hemos creado en este ejercicio y que están en nuestro espacio de trabajo con ls y guardarlos para usarlos en el futuro con save.image.
ls()
save.image("MNP.RData")Para cargarlo en el futuro tendremos que hacer load("MNP.RData"). Volveremos a estos datos para hacer otros ejercicios y aprender otros paquetes. También es trivial cambiar el código para cargar otros datos de la misma página: por provincias, municipios, datos económicos, sociales, etc.
Código completo
# Cargar librerías y datos
##########################
library(dplyr) # Manipulación de data.frames
library(tidyr) # Datos ordenados
library(readxl) # Leer ficheros excel
library(lattice) # Gráficos
download.file("http://internotes.cajaespana.es/pubweb/decyle.nsf/E27E3D29593A8FC1C12578720023B795/$File/Regiones_Demografia_MNP.xls",
destfile = "Regiones_Demografia_MNP.xls", mode="wb")
MNPorig = read_excel("Regiones_Demografia_MNP.xls",skip=10)
# Transformar datos: Datos ordenados
#####################################
MNPlargo = MNPorig %>% gather(columna,valor,-1,-2)
Encoding(MNPlargo$columna) = "UTF-8"
MNPlargo = MNPlargo %>%
separate(columna,c("Variable","Fecha"),sep=" 2",convert=TRUE) %>%
mutate(Fecha = Fecha+2000)
MNP = MNPlargo %>% spread(Variable,valor)
MNP
# Gráficos lattice
##################
xyplot(log(Nacimientos)+log(Defunciones)~Fecha|Territorio,
data=MNP,auto.key=list(space="bottom"),type=c("p","l","g"),
par.strip.text=list(cex=0.6))
xyplot(`Tasa Bruta de Natalidad (‰)` + `Tasa Bruta de Mortalidad (‰)` ~ Fecha | Territorio,
data=MNP,auto.key=list(space="bottom"),type=c("p","l","g"),
par.strip.text=list(cex=0.6))Hemos visto:
Paquetes en R:
install.packages: Instalar paqueteslibrary: Cargar paquetes
Espacio de trabajo en R:
search: Listar las carpetas de busquedals: Listar objetos en el espacio de trabajosave.image: Guardar el espacio de trabajo en un ficheroload: Cargar un espacio de trabajo en memoriaread_excel: Cargar un fichero excel (del paquetereadxl)
Información sobre objetos:
View: Abre una ventana para visualizar un objeto. Indicado con los objetos grandes. Cuidado: ¡La primera “V” es mayúscula!str: Estructura del objetoargs: Listar los argumentos de una funciónhelpò?: Abrir una página de ayuda sobre una funciónnames: Nombres de las variablesdim: Dimensiones de matrices odata.frameshead: Mostrar el comienzo de un objeto. La equivalente para el final del objeto es la funcióntailunique: Lista los valores distintos que toma un objeto (generalmente una columna ó un objeto simple)
Transformación de datos:
Encoding: Especificar el formato de codificación de un vector de caracteres.- Manipulación de datos (
tidyr): gather: Pasar de forma ancha a largaspread: Pasar de forma larga a anchaseparate: Separar una columna en varias
Manipulación de datos (dplyr):
%>%: Operador después o entonces.CTRL+MAYUS+Men RStudiomutate: Transformar o crear nuevas variables en undata.frame
Gráficos (lattice)
- Formulas en R,
y ~ x | z xyplot- Argumentos
auto.key,type,par.strip.text
Referencias básicas
- Cómo instalar R y RStudio (Universidad de Granada) http://wdb.ugr.es/~bioestad/guia-r-studio/practica-1-r-studio/
- ImpatientR (Patrick Burns) http://www.burns-stat.com/documents/tutorials/impatient-r/
- Introducción a
dplyr(José R. Berrendero) http://rpubs.com/joser/dplyr (parte de un conjunto de recursos sobre R disponible en https://caminosaleatorios.wordpress.com/el-entorno-r/) - Data Processing with dplyr & tidyr (Brad Boehmke) https://rpubs.com/bradleyboehmke/data_wrangling
- Using Lattice Graphic in R (Harry Erwin) https://fas-web.sunderland.ac.uk/~cs0her/Statistics/UsingLatticeGraphicsInR.htm
Chuletas de manipulación de datos en R (R Studio)
Referencias generales sobre R
CURSO FUNDAMENTOS ESTADÍSTICOS DE INVESTIGACIÓN. INTRODUCCIÓN A R (Universidad de Murcia) http://www.um.es/ae/FEIR/10/
Referencias citadas
[1] S. M. Bache and H. Wickham. magrittr: A Forward-Pipe Operator for R. R package version 1.5. 2016.
[2] H. Wickham. “Tidy Data”. In: Journal of Statistical Software 59.10 (2014). DOI: 10.18637/jss.v059.i10. <URL:http://dx.doi.org/10.18637/jss.v059.i10>.
Notas
- Para escribir la tilde en el teclado español tenéis que escribir la combinación ALT-GR+4 seguida de ESPACIO.↩
- Para escribir la barra vertical tenéis que escribir ALT-GR+1 seguido de ESPACIO.↩
- Existen paquetes alternativos para realizar gráficos en R. Los gráficos base, incluidos en el entorno básico, se basan en la función
plot. El paquetelatticedispone de muchos gráficos que se definen utilizando una fórmula. El otro gran sistema de gráficos, el más flexible de todos aunque un poco más complejo de comenzar a utilizar, esggplot2. Existen otros paquetes complementarios que ayudan a hacer gráficos con estos tres sistemas, y otros sistemas alternativos con posibilidades más avanzadas como gráficos dinámicos o interactivos. Más información en http://oscarperpinan.github.io/intro/graficos.html↩


Hola,
muchas gracias por este tutorial. Una cosa, el enlace para descargar los datos no funciona. ¿Sabes si se pueden descargar desde otro enlace para poder practicar?
Un saludo,
Hola, Gonzalo.
Me alegro que te sea útil. Efectivamente la página web dejó de estar operativa, pero se pueden seguir bajando los datos de archive.org. No lo he actualizado aquí, pero tienes este ejemplo actualizado en el enlace http://rpubs.com/jaortega/153198 y un tutorial más ampliado en el enlace
http://rpubs.com/jaortega/tidyverse2